L’important développement des technologies de séquençages haut-débit et des méthodes de code-barres ADN appliquées à l’ADN environnemental (ADNe) offrent de nouveaux outils pour évaluer la diversité et structuration des réseaux écologiques avec un éclairage à la fois qualitatif et quantitatif (nature et force des interactions).
Le protocole DNAquatroph permet d’analyser l’ADNe digestif (fèces, pelotes, contenus stomacaux) de plusieurs dizaines à centaines d’échantillons simultanément, sur des groupes de niveaux trophiques et/ou avec de régimes alimentaires différents.
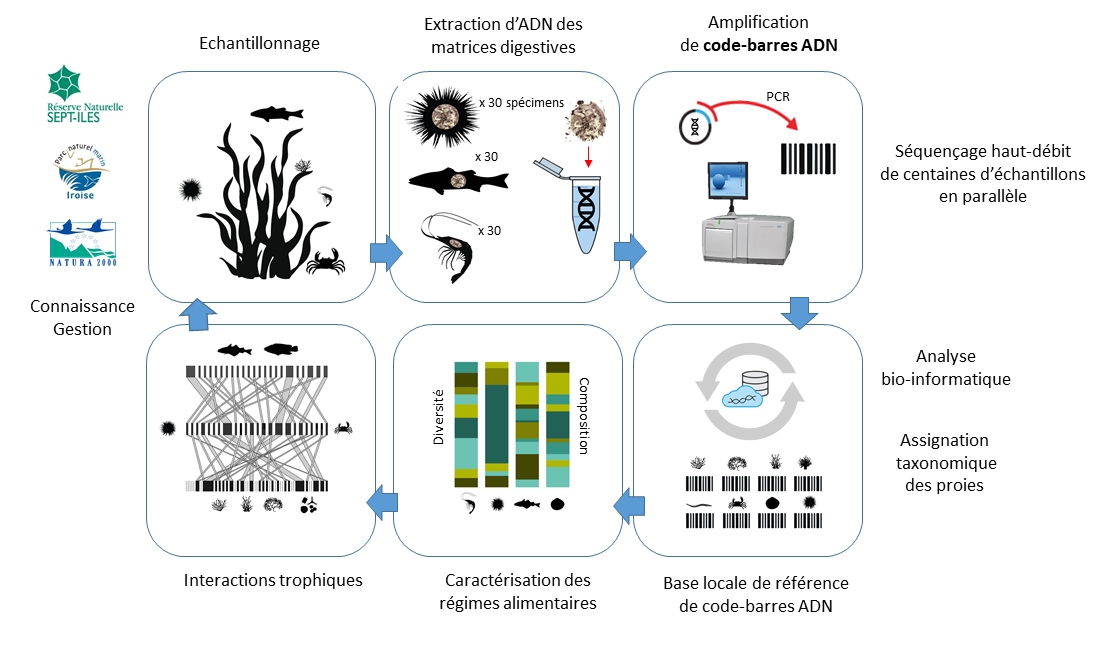
Après dissection, les échantillons digestifs sont broyés et homogénéisés au Fastprep (par microbilles) puis les ADNs sont extraits avec un kit d’extraction en plaque, avec une première étape de lyse en tubes pour minimiser les risques de contamination entre échantillons. L’analyse métabarcoding se fait en deux étapes : une première étape de PCR qui cible un marqueur ribosomal ou mitochondrial (COI, ARNr 18S, ARNr 23S) et une seconde étape de PCR qui permet d’ajouter une combinaison unique indexes à chaque échantillon, ce qui permet de multiplexer plusieurs centaines d’échantillons sur un même run de séquençage. L’analyse multi-marqueurs permet d’avoir une portrait complet des régimes alimentaires (identification des poissons, invertébrés, algues). Pour s’assurer de la fiabilité des résultats, chaque échantillon est analysé en triplicat et des témoins négatifs sont réalisés afin de contrôler les risques potentiels de contamination à chaque étape (dissection, extraction, d’amplification, séquençage).
L’approche ADNe vient compléter et préciser les résultats obtenus précédemment grâce à l’approche isotopique en identifiant précisément (à l’espèce ou au genre) les espèces impliquées dans les interactions trophiques entre les sources primaires de carbone à la base des réseaux (espèces d’algues, phytoplancton), les consommateurs primaires (brouteurs, détritivores, suspensivores) et enfin les prédateurs de ses consommateurs. Les fréquences d’occurrence (FO) et/ou la quantité moyenne de séquences (RRA) de chaque proie permet d’évaluer l’intensité des interactions.